Búsqueda de los exones con el mayor número de SNPs
Unión (‘joining’) de exones y SNPs
Recordemos que nuestro objetivo es encontrar los exones con un mayor número de SNPs. El primer paso para ello es obtener la unión (joining) de exones y SNPs (obteniendo así un listado de los exones y SNPs que solapan).
En el menú de la izquierda -> “Operate on Genomics Intervals -> Join“:
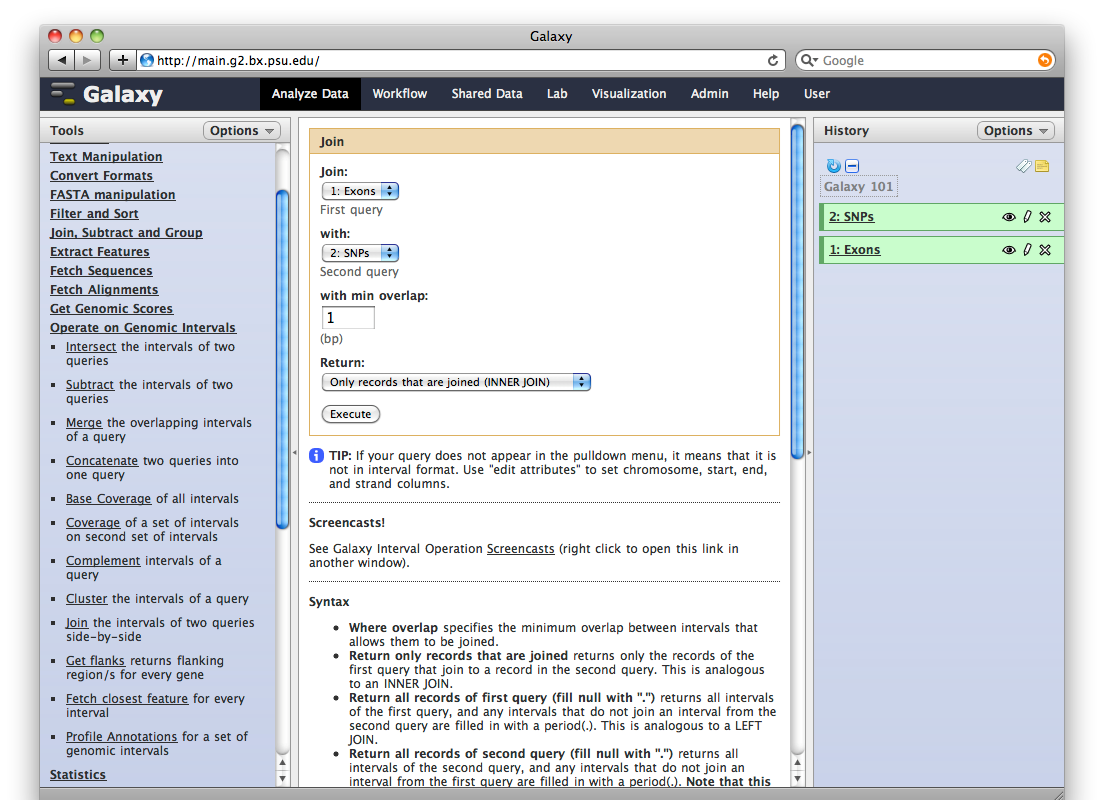 Es importante seleccionar primero el archivo más pequeño (los exones) y luego el más grande (los SNPs).
Es importante seleccionar primero el archivo más pequeño (los exones) y luego el más grande (los SNPs).
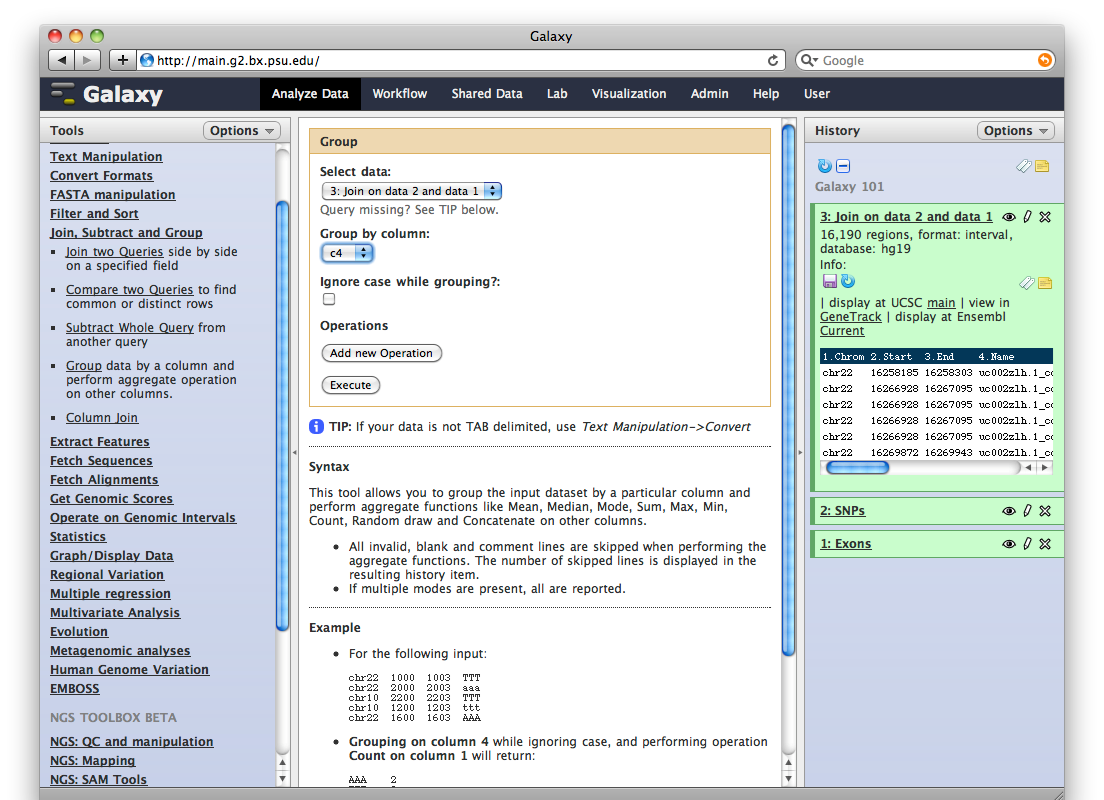
Deje las demás opciones por defecto y pinche en Execute. Obtendrá un tercer item en su historia:
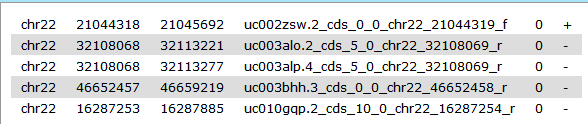
Pinchando en el botón ‘Ojo’ puede examinar los datos que contiene este nuevo item de su historia:
chr22 16258185 16258303 uc002zlh.1_cds_1_0_chr22_16258186_r 0 – chr22 16258278 16258279 rs2845178 0 +
chr22 16266928 16267095 uc002zlh.1_cds_2_0_chr22_16266929_r 0 – chr22 16267011 16267012 rs7290262 0 +
chr22 16266928 16267095 uc002zlh.1_cds_2_0_chr22_16266929_r 0 – chr22 16266963 16266964 rs10154680 0 +
chr22 16266928 16267095 uc002zlh.1_cds_2_0_chr22_16266929_r 0 – chr22 16267037 16267038 rs2818572 0 +
chr22 16266928 16267095 uc002zlh.1_cds_2_0_chr22_16266929_r 0 – chr22 16267031 16267032 rs7292200 0 + ...
Observe detenidamente estos datos (tenga en cuenta que pueden variar ligeramente si está haciendo este ejercicio con una versión más reciente de la base de datos).
Las primeras seis columnas corresponden a los exones y las seis últimas a los SNPs.
Puede ver, por ejemplo, que el exon con ID uc002zlh.1_cds_2_0_chr22_16266929_r contiene cuatro SNPs con IDs rs7290262, rs10154680, rs2818572, y rs7292200.
Número de SNPs por exon
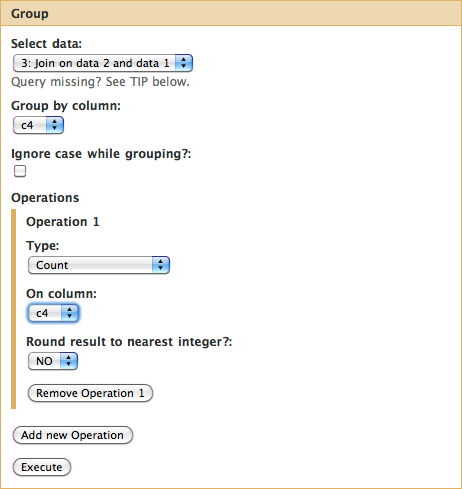
Hemos visto que el exon uc002zlh.1_cds_2_0_chr22_16266929_r se repite cuatro veces. Por tanto, podemos calcular el número de SNPs por exon contando simplemente el numero de repeticiones del nombre de cada exon. “Join, Subtract, and Group -> Group“:
Seleccione la columna 4 escribiendo “c4″ en Group by column. Pinche luego en Add new Operation y asegúrese de que las opciones que elija sean exactamente las que se muestran a continuación:
Pinchando en Execute su historia aparecerá ahora como sigue:
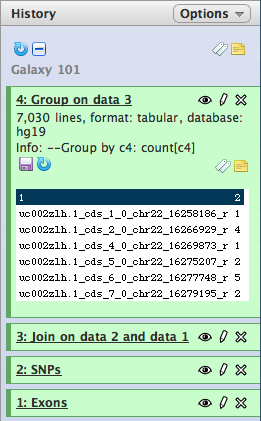
Los resultados contienen dos columnas. La primera es el nombre del exon, mientras que la segunda indica el número de veces que se repite (que equivale al número de SNPs que contiene cada exon).
Ordenando la lista por el número de repeticiones
Para determinar los exones con más SNPs, debemos editar esta lista ordenando por la segunda columna en orden descendente. “Filter and Sort -> Sort“:
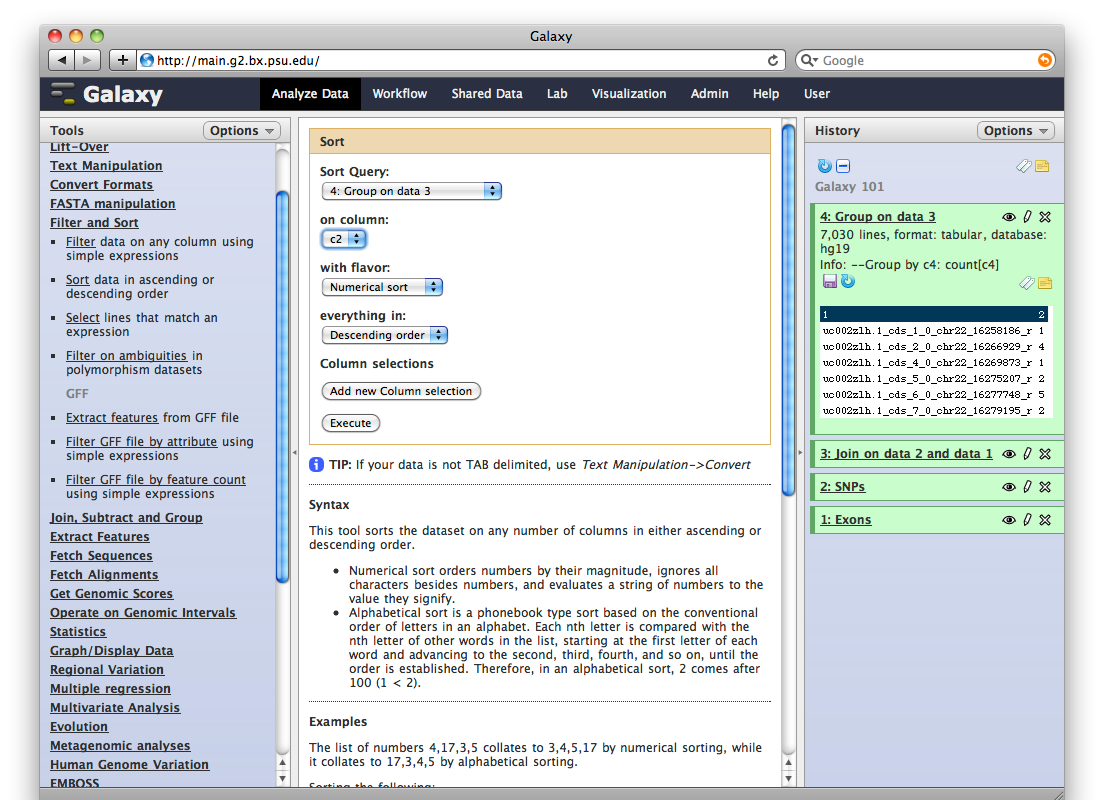
Esto dará lugar al quinto item en su historia:
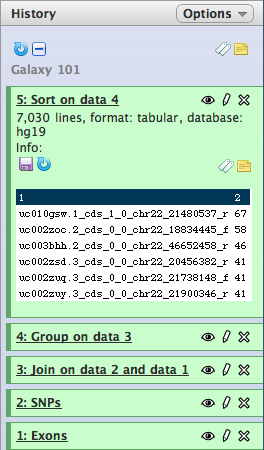
con lo que puede ver que el numero mayor de SNPs por exon es 67 (este número puede variar si esta usando una versión más reciente de la base de datos).
Selección de los cinco primeros
“Text Manipulation -> Select First“:
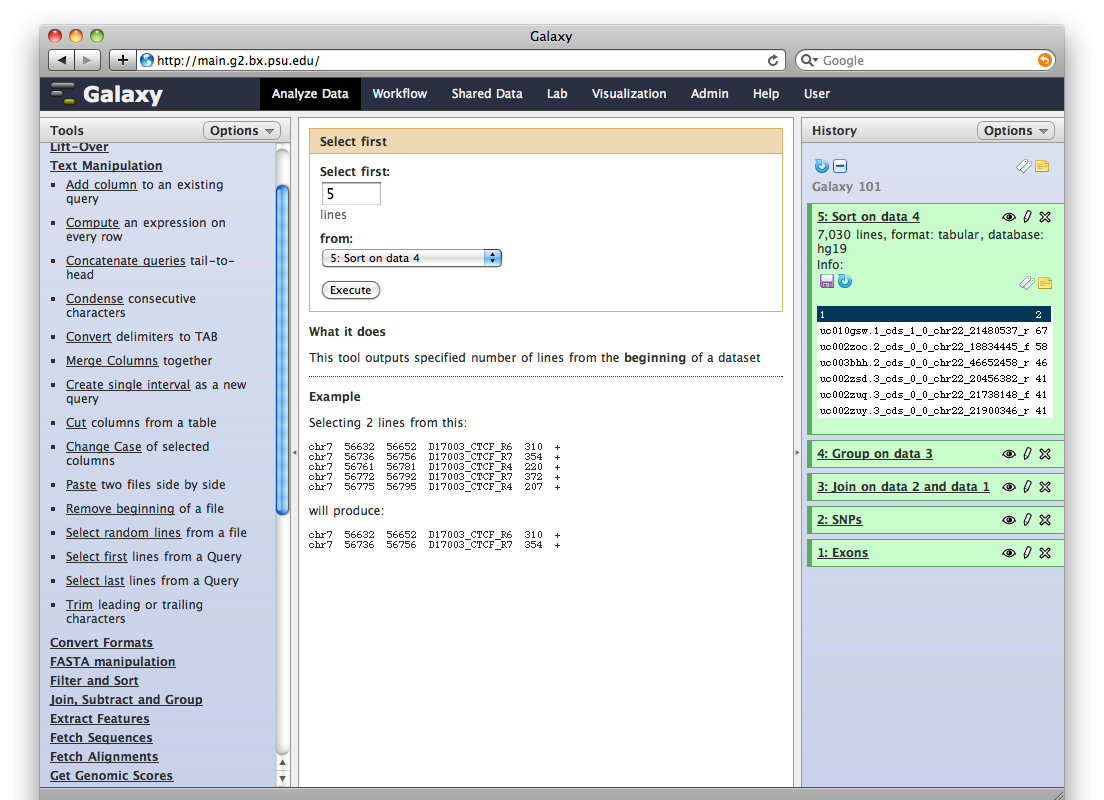
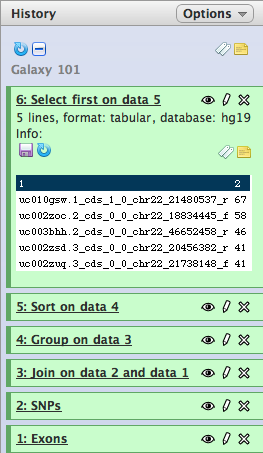
Pinchando en Execute se generará el sexto item en su historia, que contendrá sólo cinco líneas:
Recuperación de las coordenadas de los exones
Ahora sabemos (item más reciente en su historia) que los cinco exones con más SNPs contienen entre 41 y 67 SNPs (de nuevo, recuerde que estos números pueden variar si esta usando una versión más reciente de la base de datos).
Para obtener más información acerca de estos exones, debemos recuperar su información posicional (sus coordenadas). Esta información se ha perdido en el proceso de agrupamiento, y ahora solo tenemos dos columnas.
Para volver a obtener las coordenadas, debemos emparejar los nombres de los exones en el último item (columna 1) con los nombres de los exones en el dataset #1 (columna 4).
“Join, Subtract and Group -> Compare two Queries” (preste atención a los valores seleccionados en cada campo del panel central):
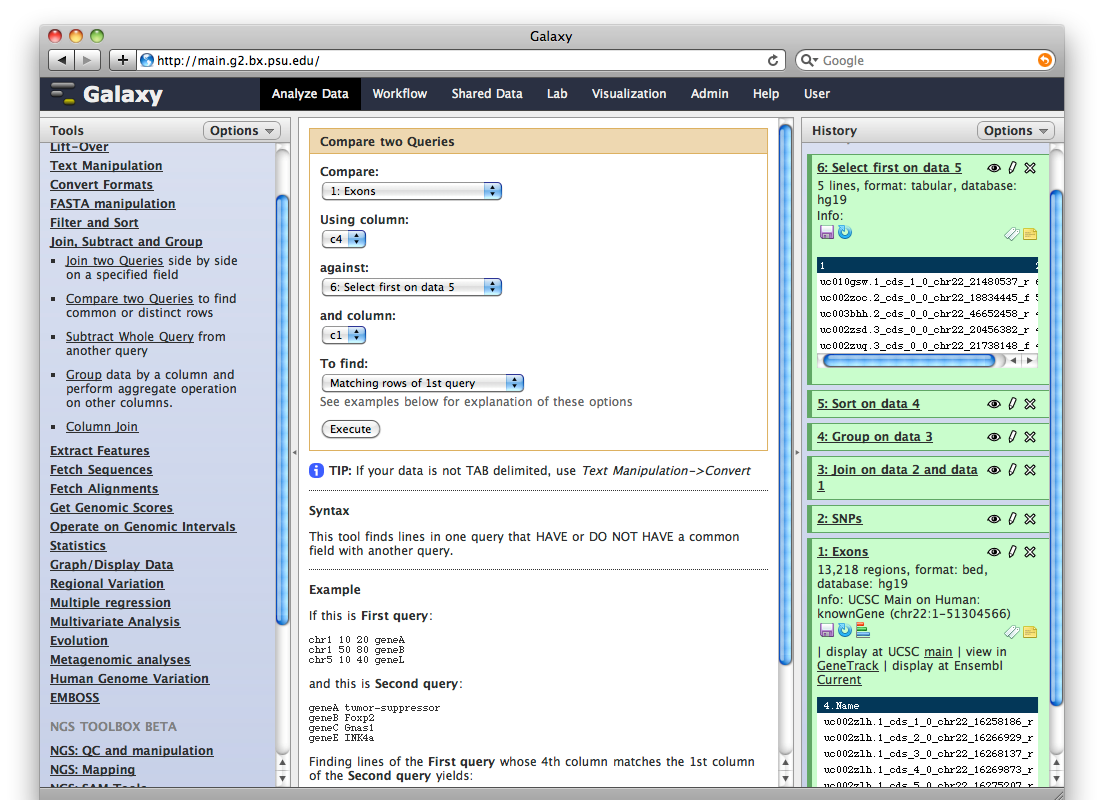
lo que añade el item 7 a la historia:
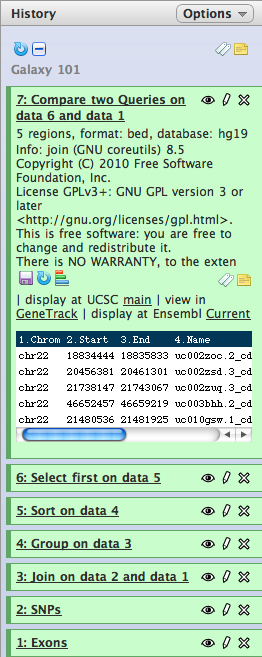
Visualización de los resultados
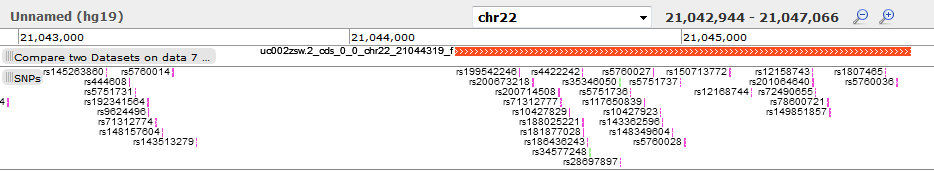
Con el botón ‘Ojo’, puede examinar los últimos datos obtenidos:
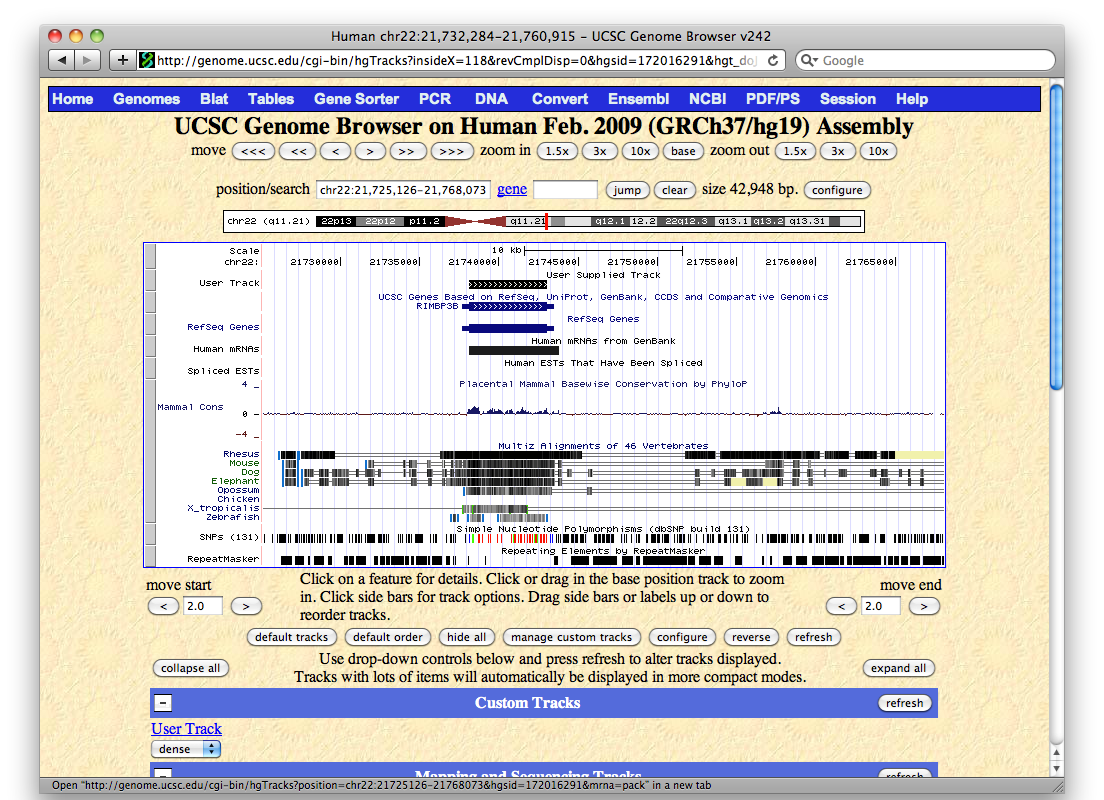
Si pasa el ratón sobre estas lineas, le aparecerá a la izquierda un icono que le permitirá visualizar el contexto genómico de cada uno de estos exones. Añada a la visualización los SNPs y podrá obtener una imagen como esta:
Puesto que ya tenemos sus coordenadas, la mejor manera de aprender más sobre estos exones es representarlos en su contexto mediante un navegador genómico. Tiene dos opciones – UCSC Genome Browser y Ensembl:

Por ejemplo, pinchando en “display at UCSC main” obtendrá una imagen parecida a esta si hace zoom sobre los distintos exones (el track con sus datos aparecerá en la parte de arriba de la imagen “User Track”):