Información básica de una secuencia
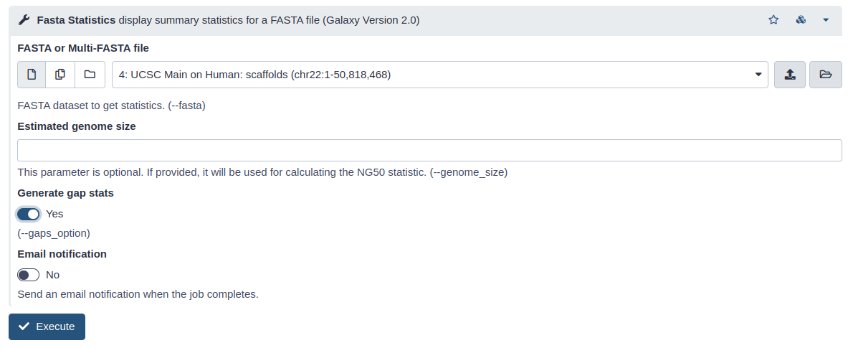
La herramienta Fasta Statistics en el menú FASTA/FASTQLas herramientas en Galaxy automáticamente detecta los conjuntos de datos que podemos usar y los muestra en un menú desplegable.
En los resultados del programa encontramos la longitud de la secuencia, el contenido G+C (incluyendo las Ns), la estadística de los bloques de Ns (gaps) y el conteo de nucleótidos.
Composición en dinucleótidos
El análisis de la composición de dinucleótidos es crucial para entender la funcionalidad y evolución del genoma, así como para identificar regiones críticas en la regulación génica y susceptibilidad a mutaciones.
compseq del paquete EMBOSS nos permite cuantificar los dinucleótidos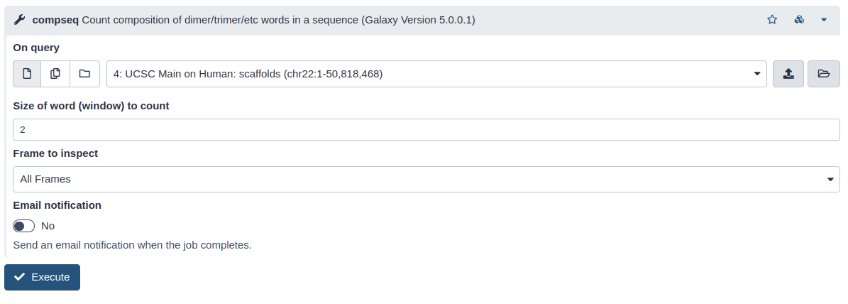
Para detectar la frecuencia de dinucleótidos se desliza una ventana móvil de tamaño 2 pbs a lo largo de la secuencia. En cada paso se anota el dinucleótido detectado.
Secuencia: ATTCCGTGAACTG
Dinucleótidos: AT, TT, TC, CG, GT…
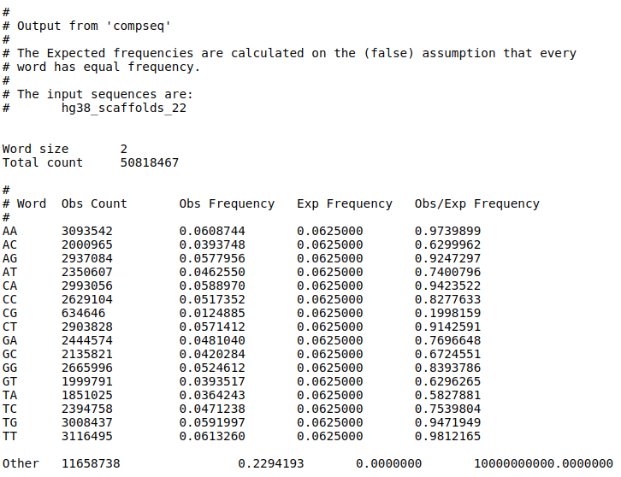
- Word: el N-mero (el dinucleótido en este caso)
- Obs Count: el número de veces que la secuencia contiene este N-mero
- Obs. Frequency: La frecuencia observada (count – ’número total de palabras’). El número de palabras existentes en este caso es ‘longitud de la secuencia’ -1.
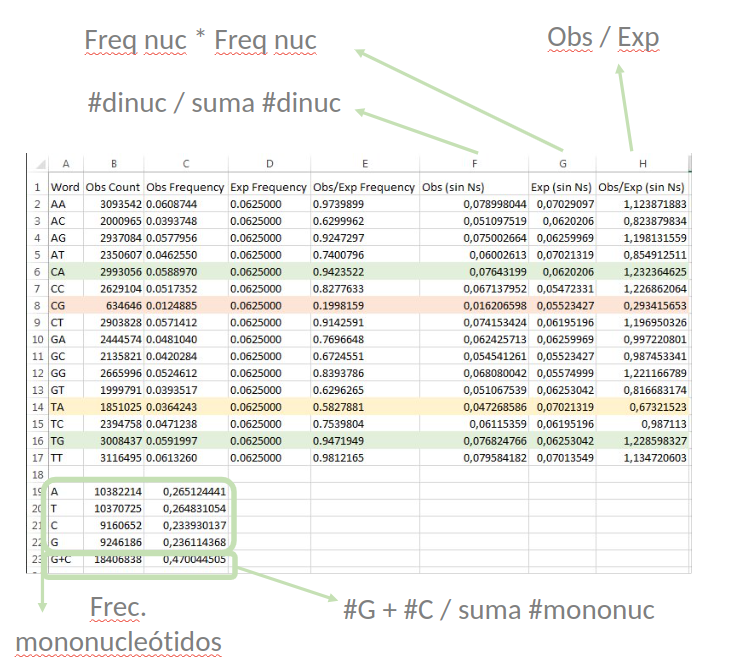
- Exp. Frequency: La frecuencia esperada según las frecuencias de los mononucleótidos
- Obs/Exp Frequency: La ratio entre la frecuencia observada y la esperada. Un valor de 1 indica que la secuencia contiene exactamente el número que hubiéramos esperado. Una desviación de 1 puede indicar la existencia de procesos biológicos que causan que observemos más o menos dinucleótidos de cierto tipo.
¿Por qué es falsa la asunción de que todas las palabras tienen la misma frecuencia esperada? ¿Cómo se calcula la frecuencias esperada correctamente? Usa valores de secuencia sin Ns para recalcular.
ANTES DE CONTINUAR COMPLETA EN LA TABLA COMPARTIDA:
La información composicional para todas tus secuencias
Busca una explicación a los valores más extremos de las distribuciones de la relación entre frecuencia observada y esperada