A diferencia de los procariotas, que se componen principalmente de ADN no-repetido (secuencias de ADN únicas en el genoma), los organismos eucariotas contienen proporciones variables de secuencias de ADN con varias copias en el genoma (elementos repetidos). En términos generales estos elementos pueden clasificarse en función de tres criterios diferentes: su número de copias, su distribución en el genoma y/o su mecanismo de propagación (duplicación o inserción).
Clasificación por frecuencia
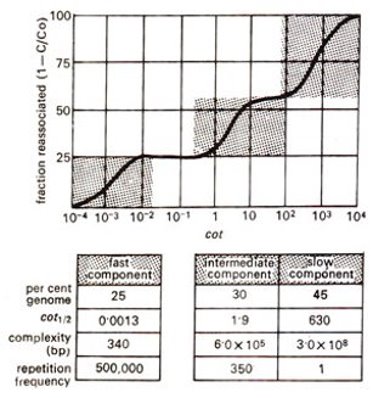
El ADN repetitivo fue descubierto en 1968 por Britten and Kohne mediante experimentos de desnaturalización y reasociación del ADN (DNA denaturation and anneling). La cinética de reasociación se mide mediante el valor C0t, donde C0 es la concentración molar inicial de las hebras complementarias de ADN, y t es el tiempo de incubación necesario para completar la mitad de la reacción de reasociación. De esta manera, secuencias de ADN con muchos fragmentos idénticos se encontrarán e hibridarán más rápido que secuencias de ADN con menor frecuencia de fragmentos repetidos.
Por lo tanto, teniendo en cuenta esta cinética de reasociación, los elementos repetidos puedes clasificarse en tres grupos:
- Altamente repetitivo (105-107 copias): secuencias con cinéticas de reasociación rápidas. Se encuentran entre un 10-15% en los genomas de mamíferos y se corresponden con secuencias repetidas en tándem.
- Moderadamente repetitivo (10-105 copias): secuencias con cinéticas de reasociación intermedias. Se encuentran entre un 25-40% en los genomas de mamíferos e incluyen la mayoría de los (retro)transposones.
- Elementos únicos o de baja repetición (1-10 copias): secuencias con cinéticas de reasociación lentas. Se encuentran entre el 40-60% en los genomas de mamíferos.
Clasificación por distribución en el genoma
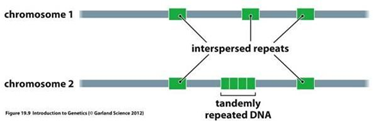
- Repeticiones en tándem, secuencias de ADN repetidas que forman grupos, principalmente ADN satélites que a su vez pueden clasificarse en múltiples grupos en función de la longitud de su motivo repetido y su localización con respecto a otros elementos genómicos. Son secuencias del genoma que pueden separarse del resto del ADN genómico mediante métodos físicos, esto se debe a que suelen contener frecuencias extremamente bajas o altas de contenido GC.
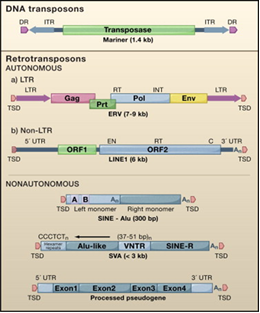
- Elementos dispersos, compuestos por diferentes tipos de elementos móviles o transponibles (TE), existen diferentes clases de estos elementos como son los SINEs, LINEs, LTRs, pseudogenes y transposones de ADN.
Clasificación por modo de propagación
Esta clasificación hace referencia a los elementos dispersos, ya que los elementos repetidos en tándem no tienen propiamente dicho un modo de propagación, sino que se duplican mediante procesos inespecíficos de amplificación del ADN, siendo el más común la replicación en círculo rodante (Rolling circle replication).
La transposición de elementos puede ser clase I y clase II. En la transposición clase II (“cortar y pegar”), los elementos cambian su posición en el genoma, pero no cambia el número de copias. Sin embargo, el aumento del número de copias puede ocurrir por otros mecanismos, como la duplicación del ADN. Por otro lado, durante este proceso de transposición no intermedian moléculas de ARN (transposones de ADN). La transposición clase I (“copiar y pegar”) se caracteriza por estar mediado por la generación de moléculas intermedias de ARN (retrotransposones), y en este caso el elemento transponible se copia, aumentando por lo tanto el número de copias en el genoma. Los dos grupos más importantes de retrotransposones son los elementos LINEs y los SINEs. Los LINEs presentan una longitud aproximada de 7kb, aunque en el genoma (a excepción de los activos) suelen encontrarse recortados. Estos elementos contienen dos ORFs, correspondiendo uno de ellos a una transcriptasa inversa, que es la que le permite transponerse de manera autónoma. LINE1 es el miembro más importante de esta familia. Los SINEs (Short interspersed repetitive elements) son secuencias cortas (< 500 nt) y no autónomas, ya que no codifican ninguna proteína. Miembros importantes son las Alus (en primates) y B1, B2 (en roedores).
Densidad de elementos repetidos
Ahora, vamos a determinar el contenido en elementos repetidos incluyendo los transposones de ADN, retrotransposones o elementos LTR. Las anotaciones las podemos encontrar en el Table Browser de UCSC en Variation and Repeats o Repeats. A lo largo del análisis utilizaremos las anotaciones de RepeatMasker.
Usaremos Get Data > UCSC Main:
- Seleccionamos nuestro ensamblado
- En groups Variation and Repeats o Repeats y track RepeatMasker
- Formato Selected fields from primary and related tables
- En la siguiente pestaña seleccionamos: genoName, genoStart, genoEnd y RepClass (o name)
Eliminamos la posible redundancia con Operate on Genomic Intervals > Merge
Y calculamos la longitud total con Summary statistics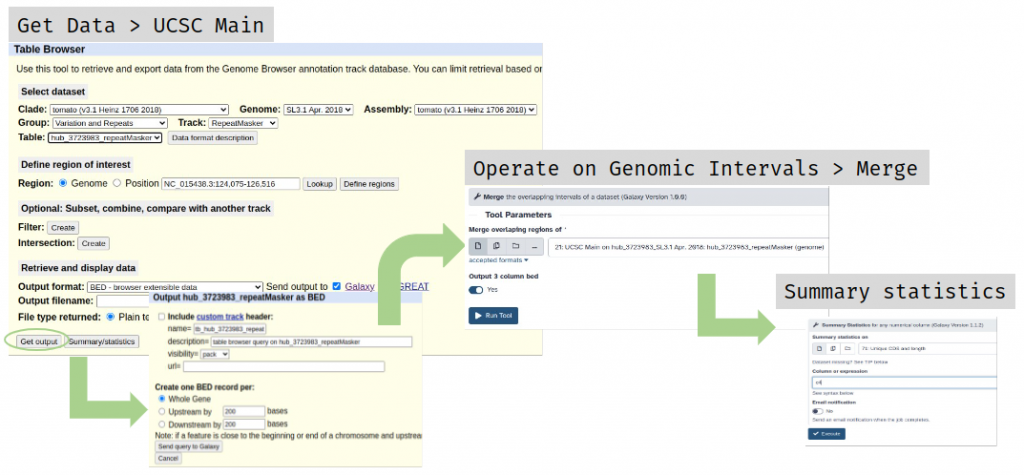
Tipos de elementos repetidos
Para cuantificar clases de elementos repetidos debemos en primer lugar separar la tabla de RepeatMasker en las diferentes clases de elementos. De entre las especies seleccionadas podemos encontrar dos tipos de tablas: las de UCSC que contienen una columna RepClass, con el nombre de la clase del elementos repetido, y las de GenArk que no lo contienen, pero podemos extraerlo de la columna name.
Tablas con RepClass
En Galaxy calculamos la longitud de todos los elementos con la herramienta Compute del menú Text Manipulation (genoEnd - genoStart)
Y mediante la herramienta Group del menú Join, Substract and Group calculamos la suma de los elementos de cada clase (agrupamos por RepClass)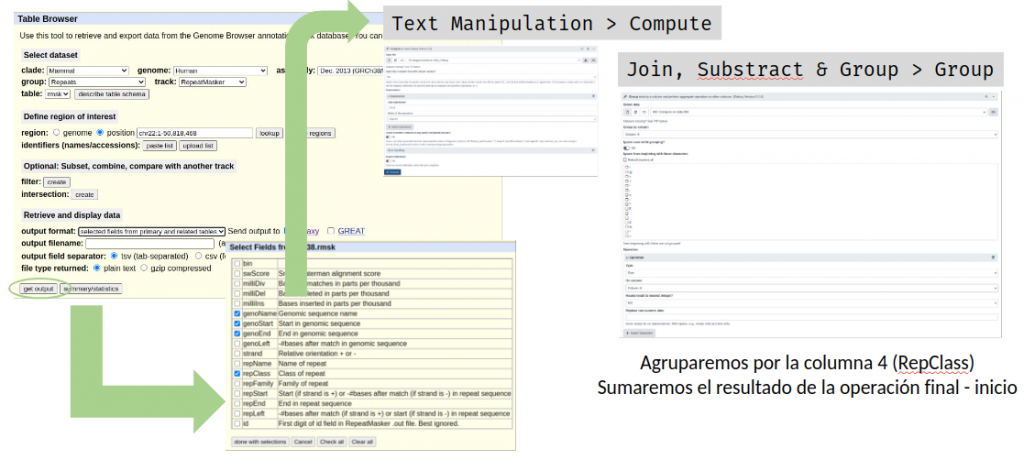
Tablas sin RepClass
En la columna name tenemos la clase y la familia del elemento separado por "#" y "/" para dividir dicha columna debemos reemplazar "#" y "/" por "," con la herramienta Replace Text in a specific column del menú Text Manipulation.
PARA REEMPLAZAR "/" es necesario utilizar el patrón "\/" en Find pattern
Después romperemos la columna usando "comma" como separador con la herramienta Cut columns from a table del menú Text Manipulation, seleccionando todas las columnas del fichero.
Ahora calcularemos las longitudes de todos los elementos con la herramienta Compute (final - inicio)
Y agruparemos por la clase del elemento y sumaremos las longitudes mediante la herramienta Group del menú Join, Substract & Group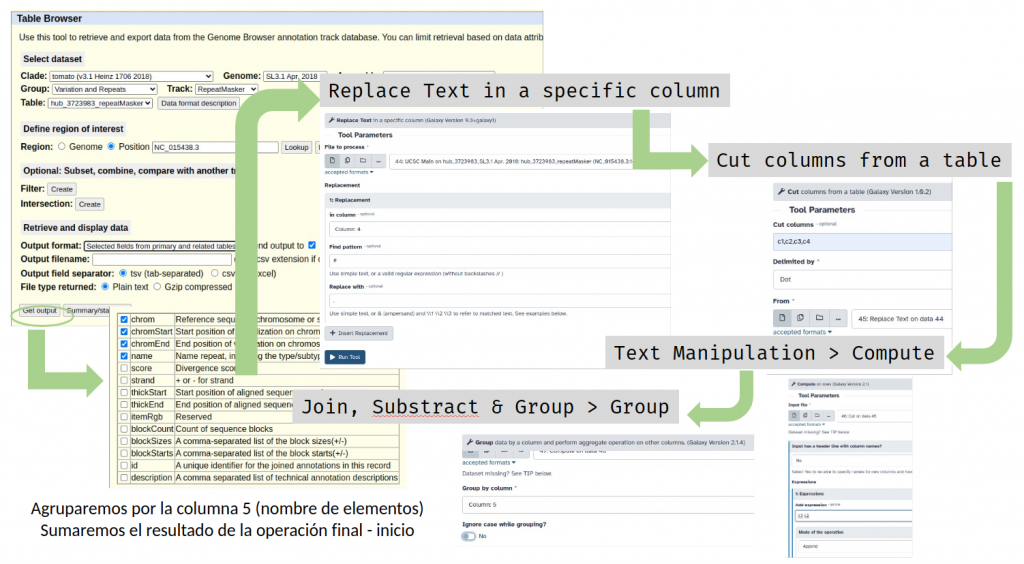
ANTES DE CONTINUAR:
Completad en la tabla compartida la densidad de elementos repetidos y de cada una de las clases