Además de la densidad génica y el tamaño del genoma, la propia estructura de los genes de un organismo puede verse moldeada por procesos evolutivos, modificando la regulación de la expresión génica y generando nuevas isoformas, las cuales contribuyen a la plasticidad fenotípica de los organismos . Las nuevas isoformas pueden aparecer por procesos en cis, generalmente mediante duplicación o exonización seguido de mutación, o en trans mediados por mutaciones en proteínas implicadas en el propio proceso del barajamiento de exones. La aparición de nuevas isoformas se ha relacionado con la adaptación, la divergencia entre especies y por supuesto con la complejidad fenotípica de los organismos. En este apartado estudiaremos la estructura de los genes, en base a sus componentes y el número de isoformas anotadas. Las anotaciones de genes las dividiremos entre genes codificantes y no-codificantes como hicimos en el apartado anterior.
Mecanismos de evolución de la estructura génica
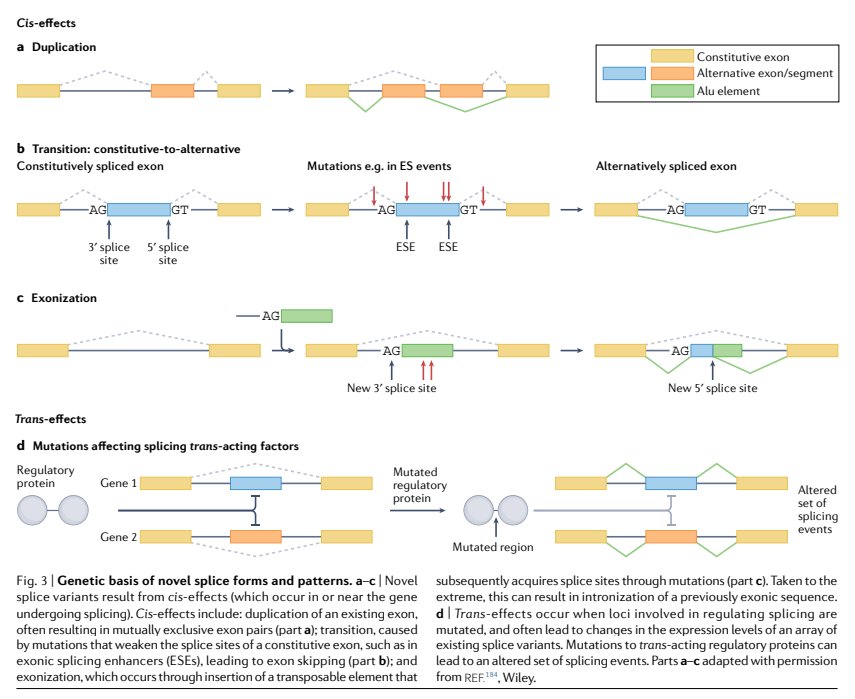
Tipo de barajamiento de exones
Número de exones por transcrito
De la misma tabla que usamos para contabilizar el número de genes por Mbp, podemos obtener el número de exones por transcrito. En este caso seleccionaríamos la casilla exonCount (en algunos casos blockCount) en la selección de campos.
Get Data > UCSC Main (Esta tabla es al misma que obtuvimos en el paso previo)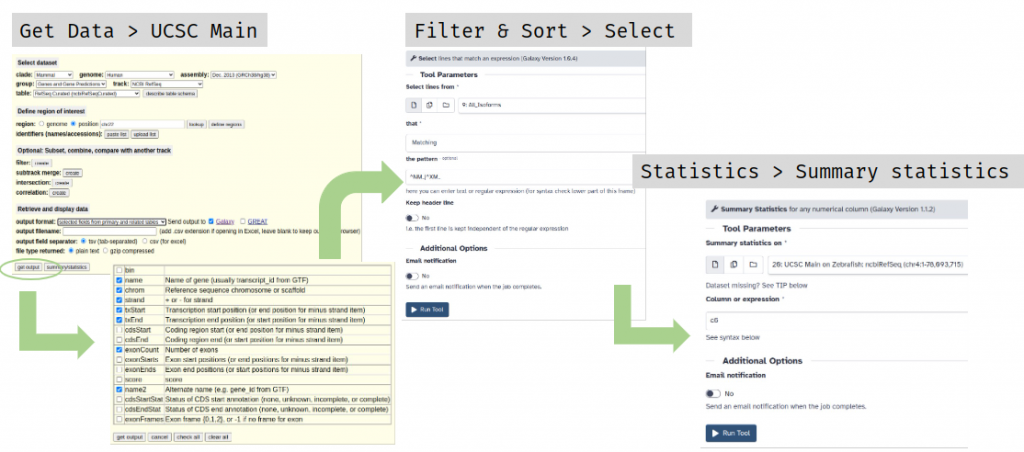
En la herramienta Select del menú Filter & Sort seleccionamos en un caso genes codificantes y en el otro no-codificantes
Y con Summary Statistics calculamos las estadísticas básicas del número de exones por transcritoLongitud de exones por transcrito
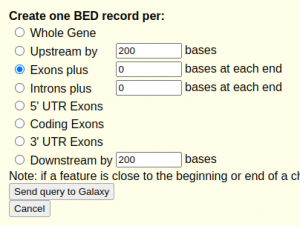
Para calcular la longitud de los exones necesitaremos obtener los exones en formato BED, por lo que procedemos como al calcular el % de nucleótidos CDS, pero en este caso en lugar de seleccionar Coding Exons, seleccionaremos Exons. Recordemos que hay que evitar las duplicidades, ya que el mismo exón puede encontrarse en diferentes transcritos (exones constitutivos). Sin embargo, en este caso no sólo nos interesa el número de pbs, sino que también nos interesan las longitudes de los exones resultantes, sitios aceptores y/o donadores alternativos (alternative 5’ and/or 3’ splice site) durante el barajamiento, ya que, aunque solapen con otros exones tienen longitudes diferentes.
Get Data > UCSC Main
- Genes en formato BED y seleccionamos Exon plusFiltramos codificantes y no-codificantes mediante Select (Filter & Sort)Para tener en cuenta el número total de exones diferentes, ya sea por tamaño o ausencia/presencia, utilizaremos la herramienta Unique para descartar aquellos exones con el mismo inicio y final que se encuentren duplicados en diferentes transcritos.
Herramienta Unique del menú Text Manipulation, en las opciones avanzadas indicamos en column start la columna con la posición de inicio de los exones y en column end el final de los exones
Una vez eliminadas las duplicidades usamos Summary Statistics sobre la longitud de los exones (final - inicio)
ANTES DE CONTINUAR COMPLETA LA TABLA COMPARTIDA CON:
Media del número de exones en genes codificantes y no-codificantes
Media de la longitud de los exones en genes codificantes y no-codificantes
Media de la longitud de intrones en genes codificantes y no-codificantes
Media del número de isoformas en genes codificantes y no-codificantes