Durante el desarrollo de estas prácticas utilizaremos tanto genomas de la base de datos de UCSC como del GenArk, proyecto que se encuentra integrado en el buscador de la UCSC. Sin embargo, para poder acceder a las anotaciones de GenArk desde Galaxy debemos siempre que empecemos a trabajar seleccionar el ensamblado de nuestra elección en el UCSC y después abrir Galaxy para continuar con nuestro trabajo.
Anotaciones de GenArk hay que cargarlas SIEMPRE en UCSC antes de abrir Galaxy
Seleccionar y cargar ensamblado de elección
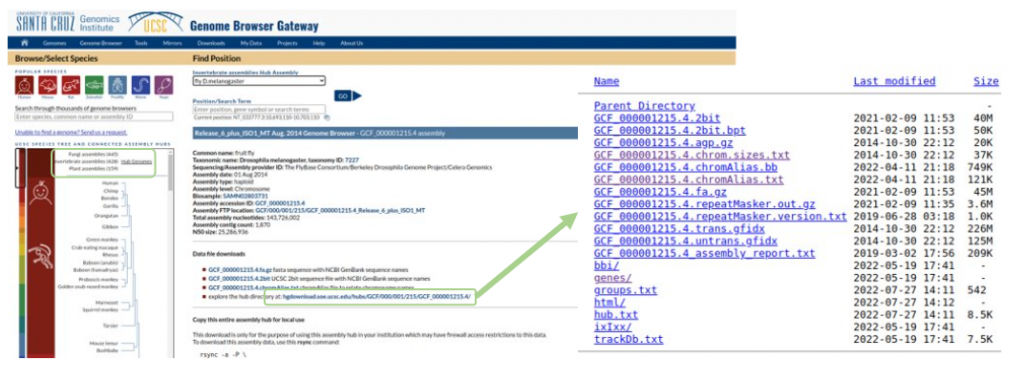
En primer lugar accederemos a la página principal del UCSC Genome Browser, donde buscaremos el genoma de referencia para la especie elegida utilizando la información proporcionada en la tabla de especies a analizar. Arriba me he referido a este paso como cargar las anotaciones.
Una vez carguemos la página de la especie en cuestión deberemos localizar 3 ficheros:
- ChromAlias: fichero tabulado que contiene los diferentes nombres que pueden recibir los cromosomas de la especie.
- ChromSizes: fichero tabulado que contiene los tamaños en nucleótidos de los cromosomas.
- *.2bit: un fichero multifasta con todas las secuencias que componen el ensamblado de la especie en formato comprimido.
En la zona inferior de la página deberíamos encontrar el enlace al repositorio HTTP o hub dependiendo de la especie, como se muestran en los ejemplos siguientes.
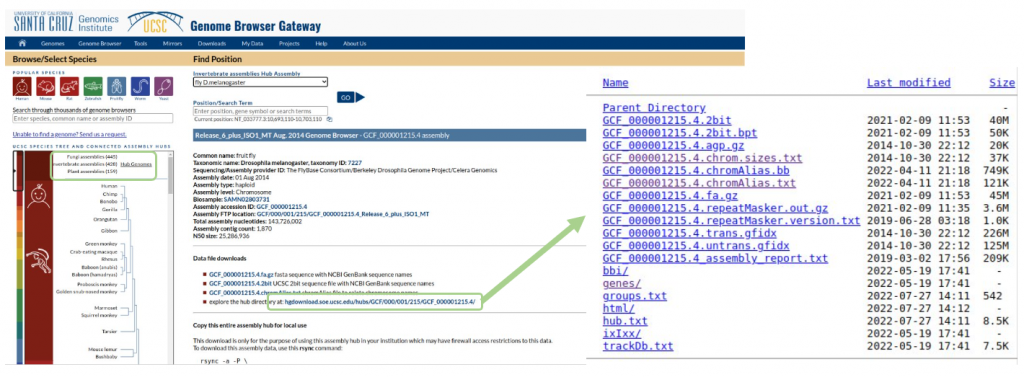
A partir de los ficheros chromSizes, chromNames y mediante una búsqueda en la red, identificaremos los cromosomas sexuales heteromórficos (si los tuviera la especie), y seleccionaremos hasta 4 cromosomas (incluyendo los sexuales) con tamaños superiores a 10 megabases, si no fuera posible se seleccionarán los cromosomas más grandes disponibles.
El fichero chromNames nos permitirá enlazar los identificadores del chromSizes con otros más informativos.
Una vez se haya completado la subida del fichero *.2bit lo descomprimiremos a ficheros fasta, en este paso extraeremos exclusivamente los cromosomas seleccionados.
Una vez seleccionados nuestros cromosomas problema, copiaremos la dirección del fichero *.2bit de la especie y lo cargaremos en nuestro historial de Galaxy.
Pulsa GET DATA en el menú de la izquierda
Y seleccionamos UPLOAD FILE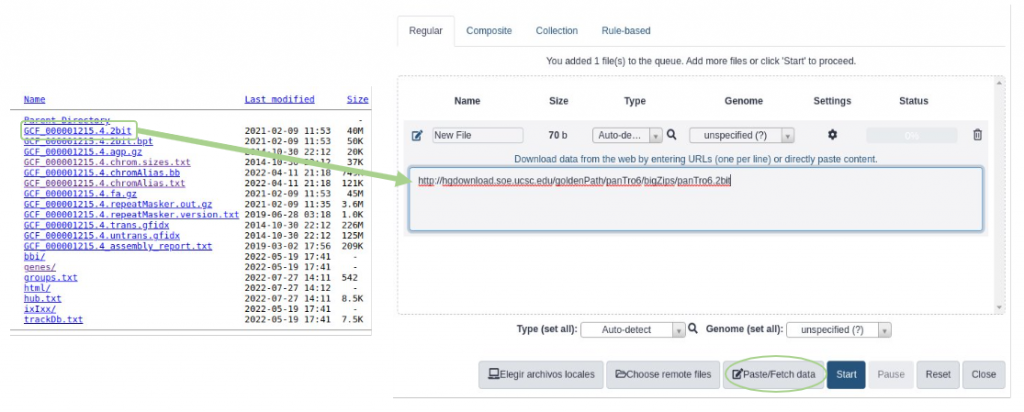
En la ventana que aparezca copiamos la dirección y seleccionamos las opciones de la imagen de arriba.
Pulsamos STARTAhora descomprimiremos uno a uno nuestros cromosomas a formato fasta.
En el menú CONVERT FORMATS selecciona la función twoBitToFa
- Opción Restrict this to just one sequence incluye el nombre de tu cromosoma
Estos pasos deberán repetirse para cada uno de los 4 cromosomas seleccionados
ANTES DE CONTINUAR COMPLETA EN LA TABLA COMPARTIDA:
Información de la especie seleccionada: Reino, Filo, Clase y Orden
Identificador más informativo de los cromosomas
Tipo de cromosoma: sexual o autosómico