Introducción a las Ontologías en Biología: GO y KEGG
Ver presentación.
Estudio del enriquecimiento en términos GO y rutas KEGG
Utilizando el paquete clusterProfiler de R, analizaremos una lista de genes cuyos productos funcionales se ubican en la mitocondria. El objetivo de este análisis es determinar si existen términos GO y rutas KEGG que estén sobre-representados en nuestra lista de genes con respecto al conjunto total de genes de esa especie (en nuestro caso, Homo sapiens).
Preparar el entorno de trabajo
Abra una terminal en el servidor de docencia y ejecute R. Ejecute las siguientes sentencias:
library(clusterProfiler)
library(org.Hs.eg.db)
library(pathview)
genes <- read.table("/home/biocomp/tema8/mitochondrial_proteins.txt", header=F)
genes <- as.character(genes$V1)
universe <- keys(org.Hs.eg.db, keys = 'ENTREZID')
Convertir de Gene Symbol a Entrez ID
genes <- bitr(genes, fromType="SYMBOL", toType="ENTREZID", OrgDb=org.Hs.eg.db) genes <- genes$ENTREZID genes <- sort(genes, decreasing = TRUE)
Obtener términos GO sobre-representados
genes_BP <- enrichGO(gene = genes,
universe = universe,
OrgDb = org.Hs.eg.db,
ont = "BP",
pAdjustMethod = "BH",
qvalueCutoff = 0.05,
readable = TRUE)
genes_MF <- enrichGO(gene = genes,
universe = universe,
OrgDb = org.Hs.eg.db,
ont = "MF",
pAdjustMethod = "BH",
qvalueCutoff = 0.05,
readable = TRUE)
genes_CC <- enrichGO(gene = genes,
universe = universe,
OrgDb = org.Hs.eg.db,
ont = "CC",
pAdjustMethod = "BH",
qvalueCutoff = 0.05,
readable = TRUE)
Obtener rutas KEGG sobre-representadas
genes_KEGG <- enrichKEGG(gene = genes,
organism = 'hsa',
pvalueCutoff = 0.05)
Visualizar los términos GO sobre-representados
pdf("genes_BP.pdf", width=10, height=10)
dotplot(genes_BP)
dev.off()
pdf("genes_MF.pdf", width=10, height=10)
dotplot(genes_MF)
dev.off()
pdf("genes_CC.pdf", width=10, height=10)
dotplot(genes_CC)
dev.off()
Transfiera los ficheros genes_BP.pdf, genes_MF.pdf y genes_CC.pdf a su ordenador para poder visualizarlos.
Visualizar las rutas KEGG sobre-representadas
pdf("genes_KEGG.pdf", width=10, height=10)
dotplot(genes_KEGG)
dev.off()
Transfiera el fichero genes_KEGG.pdf a su ordenador para poder visualizarlo.
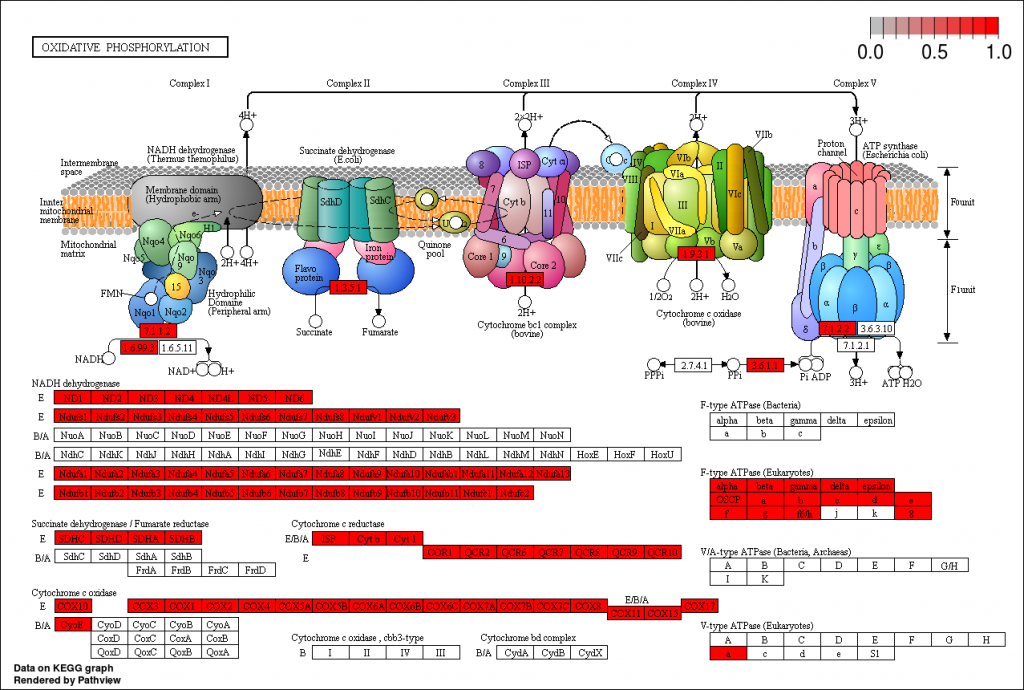
Para visualizar una ruta concreta (por ejemplo, hsa00190) ejecute:
pathview(gene.data = genes,
pathway.id = "hsa00190",
species = "hsa")
Transfiera el fichero hsa00190.pathview.png a su ordenador para poder visualizarlo.