Tenemos el fichero MGDB-pri.fa dentro de la carpeta (/opt/sRNAtoolboxDB/libs) que contiene todas las secuencias precursoras de microRNAs anotadas en la base de datos MirGeneDB.
1) reportar la longitud de la secuencia más corta, más larga y la longitud media
2) determinar la distribución del contenido en G+C. Ánalizar visualmente la distribución
3) determinar el número de especies diferentes. En el nombre de un microRNA como Aae-Mir-2-P3_pri:
- Aae es la especie – Aedes aegypti en este caso,
- Mir-2 es el nombre de la familia,
- Mir-2-P3: es el nombre del gen, con P se indica de que copia paráloga se trata
4) determinar el número de especies diferentes en las que se ecuentran un gene. Generar una salida ordenado de mayor a menor número.
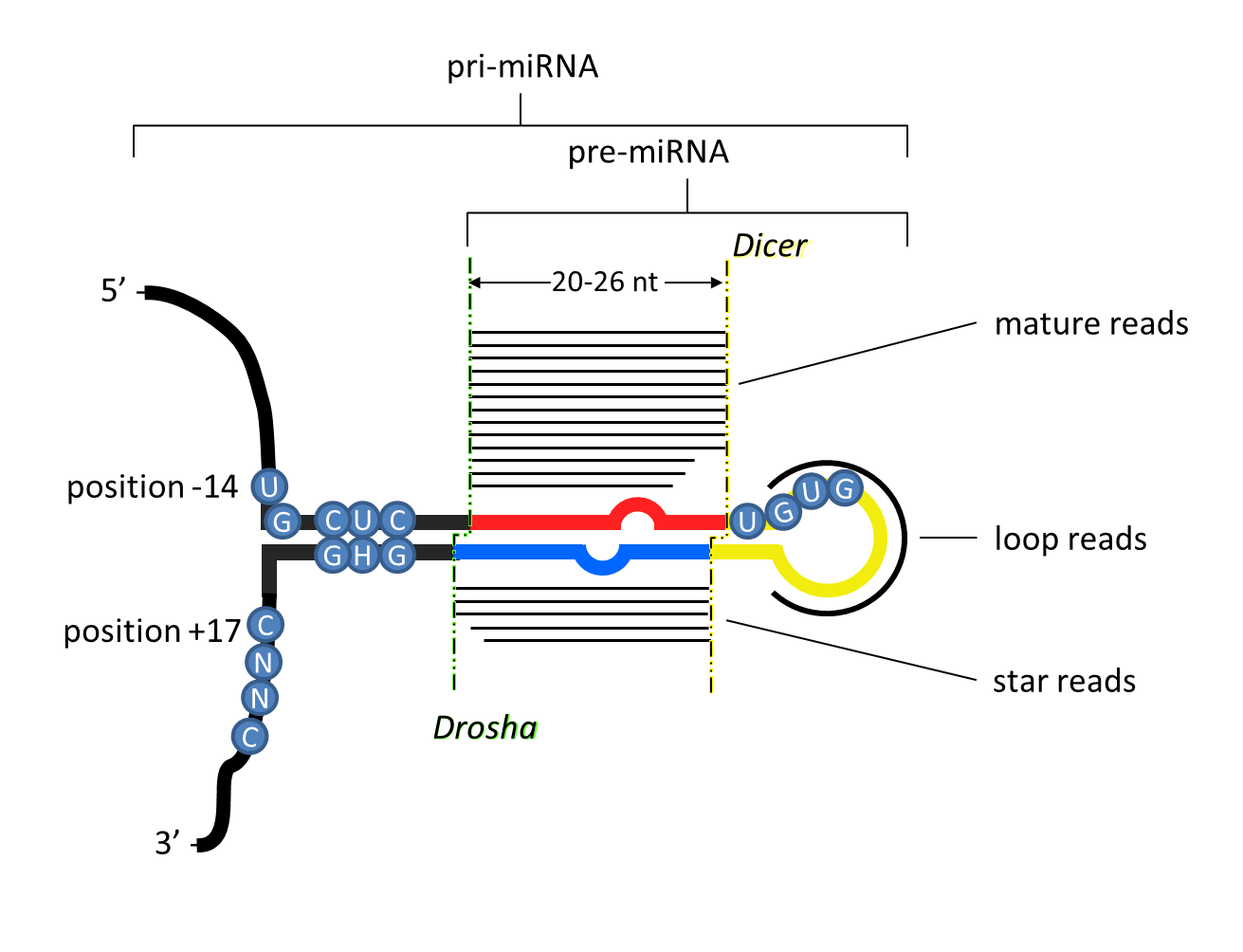
5) determinar el número de microRNA genes que contienen el patrón GNNG en la región flanqueante 3′ (véase abajo un esquema con los patrones que pueden existir en la secuencia)