Duración: 1,5 horas
Medios: Podemos usar todo MENOS al vecino, el móvil, programas de chateo u otras formas de comunicar con terceros
Las respuestas se dan en un fichero (txt, word o PDF) que se mandará a al finalizar la prueba poniendo ‘prueba Genómica Funcional’ en el asunto. Si procede, mandar también las hojas de cálculo.
Tanto el nombre del fichero como el fichero como tal deberían indicar el nombre del alumno.
IMPORTANTE: ¡Razonar brevemente todas las respuestas!
Trazar las soluciones mediante capturas de pantalla.
1) (5 puntos) Tenemos la siguiente entrada en la base de datos miRBase: MI0040629
Discutir si se puede tratar de un verdadero microRNA o un falso positivo.
2) (8 puntos) Tenemos este fichero multifasta que contiene las secuencias precursoras (hairpin) del microRNA x4 en 5 especies diferentes.
- Determinar el brazo funcional
- Indicar la posición (marcando la posición en una captura de pantalla o mediante coordenadas) más probable del microRNA funcional dentro de la secuencia humana
3) (11 puntos) Se sospecha que el microRNA hsa-miR-34a-5p podría regular al gen MYCN
- (3 puntos) Reportar la secuencia de hsa-miR-34a-5p y confirmar que se trata del brazo funcional (es suficiente reportar una línea de evidencia)
- (2 puntos) Demostrar (mediante el uso de una base de datos) que existe evidencia experimental que avala esta interacción
- En un paciente con cáncer se ha encontrado el hsa-miR-34a-5p mutado. En este fichero tenemos 4 posibles regiones de unión frente a la 3’UTR de MYCN, dos para la secuencia canónica y dos para la mutada.
- (2 puntos) ¿Que cambios en la estructura del duplex y energía libre podemos observar?
- (2 puntos) ¿Que impacto puede tener la mutación en la regulación del gen MYCN?
- (2 puntos) ¿Que consecuencia puede tener la pérdida de regulación de miR-34a-5p sobre MYCN si tenemos en cuenta que éste es un importante proto-oncogen?
4) (12 puntos) Se ha secuenciado un total de 7 muestras de la garrapata Ixodes scapularis de dos tejidos diferentes, 4 del tejido_1 y 3 del tejido_2. Hemos obtenido la siguiente matriz que contiene el número total de lecturas asignadas a cada microRNA en las diferentes muestras.
- (4 puntos) Normalizar los valores de la matriz (RPM, lecturas por million)
- (2 puntos) ¿Existen muestras con un perfíl de expresión muy diferente, es decir que podrían haber sufrido algún tipo de problema (extracción de muestra, generación de librería, secuenciación, etc)?
- (2 puntos ) Agrupar las muestras y cuando es posible asignar cada una a un tejido
- (4 puntos) Calcular los foldchanges (FC, magnitud de cambio) entre las muestras S1+S3 y S4 + S5. ¿Cuantos microRNAs cambian sus niveles de expresión al menos un factor 4 entre S1+S3 y S4+S5?
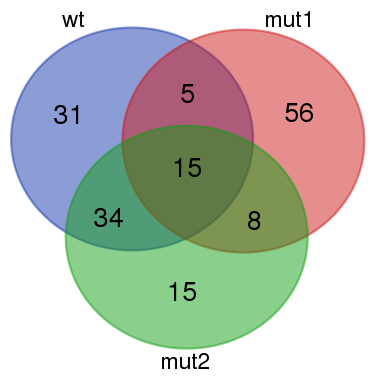
5) (4 puntos) Tenemos 3 secuencias de microRNAs; la secuencia ‘wild type’ y dos que contienen una mutación cada una. Después de detectar los genes dianas de cada una de las 3 secuencias, se representan los conjuntos de genes en forma de un diagrama Venn (véase abajo). ¿Cuál de las dos secuencias mutadas tendrá la mutación dentro de la región semilla? Razonar brevemente.