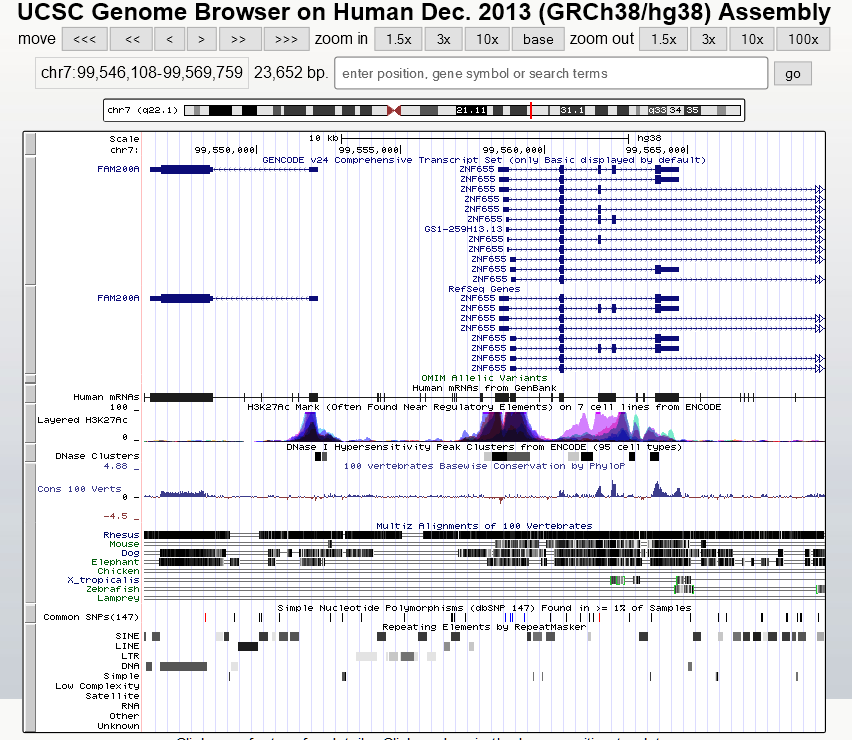
Los navegadores genómicos permiten explorar genomas completos, mostrando las diferentes anotaciones en pistas apiladas unas sobre otras. Esto permite resumir de manera gráfica una ingente cantidad de información sobre una determinada región del genoma.
UCSC Genome Browser
Algunos genes interesantes
- El color de ojos: OCA2
- El color rojo del pelo: MCR1
- Fibrosis quística: CFTR
- Cáncer de mama: BRCA1, BRCA2
- Intolerancia a la lactosa: MCM6
- Sociabilidad y empatía: OXT
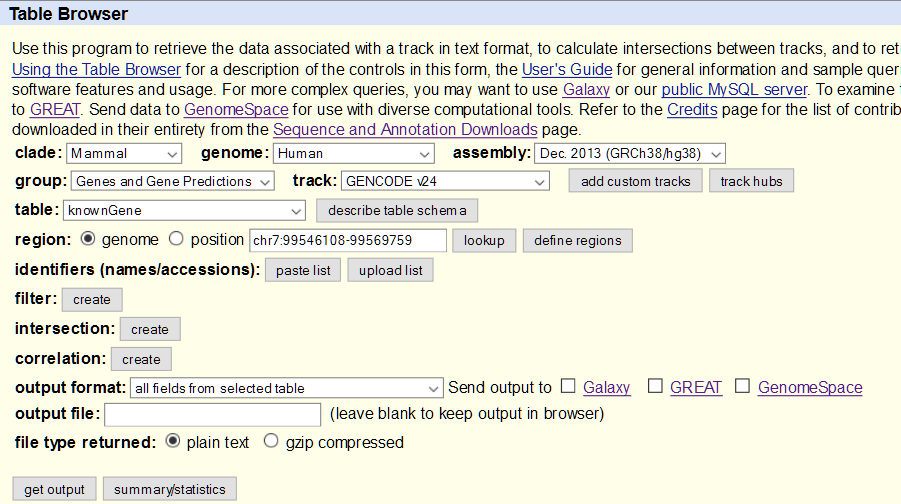
Table Browser
Permite descargar datos genómicos en diferentes formatos: bed, gff/gtf, fasta, etc. Los datos a los que podemos acceder mediante el Table Browser son los mismos que podemos visualizar en el Navegador Genómico.
El formato BED
Coordenadas UCSC
Operaciones sencillas con Table Browser
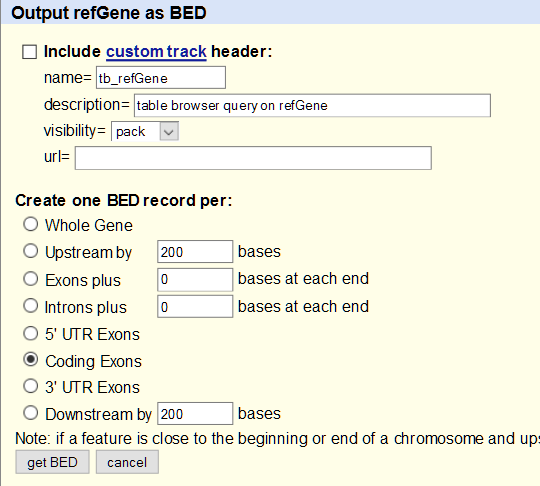
Obtener los exones de los genes RefSeq del cromosoma 21 humano
Tras pulsar ‘getBED’ obtendrá la tabla:
chr21 5026479 5026630 NM_001283052_cds_2_0_chr21_5026480_f 0 +
chr21 5027934 5028225 NM_001283052_cds_3_0_chr21_5027935_f 0 +
chr21 5032052 5032217 NM_001283052_cds_4_0_chr21_5032053_f 0 +
---
Puede guardar la tabla a disco (desde el menú Archivo del navegador -> Guardar como: ‘chr21_exons.txt’) e importarla en una hoja de cálculo para obtener una estadística básica de la longitud de los exones del cromosoma 21 (longitud media, mínima y máxima, desviación standard, histograma de frecuencias, etc.).
Si repite la operación con los intrones puede hacer un estudio comparado de las longitudes de exones e intrones.
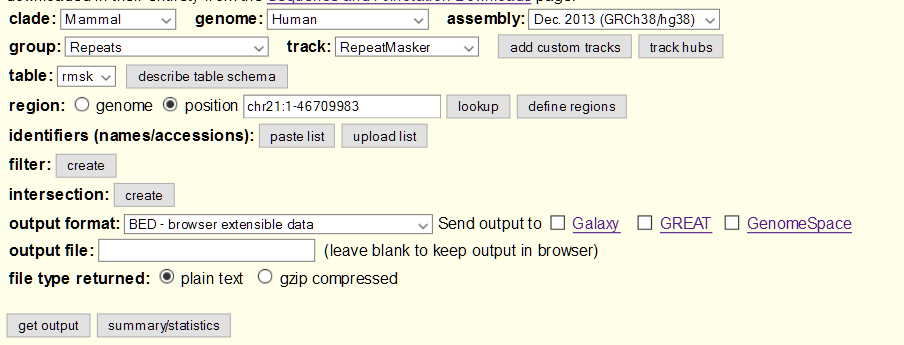
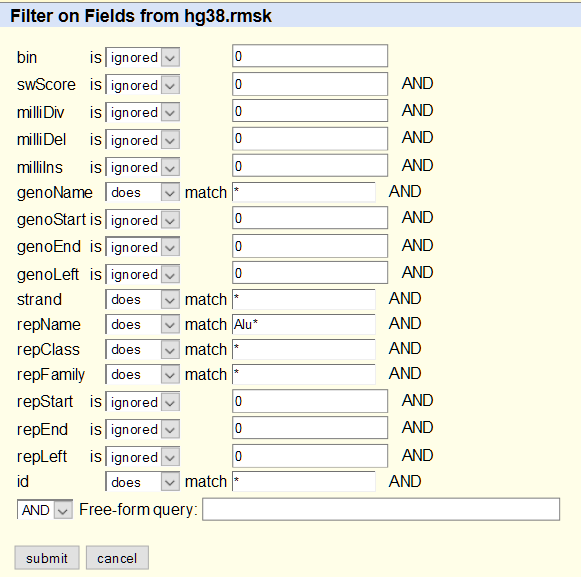
Uso de filtros: Obtener los retrotransposones de la familia Alu en el cromosoma 21 humano
Pinche en filter -> create:
Tras presionar sobre ‘Submit’ -> ‘Get output’ -> get BED obtendrá la tabla con las Alus del cromosoma 21 humano:
chr21 5010000 5010061 AluYk11 465 -
chr21 5010563 5010892 AluY 2392 -
chr21 5010955 5011213 AluJb 1590 +
---
Si importa esta tabla en una hoja de cálculo, puede obtener una estadística básica de las longitudes de los retrotransposones Alu en el cromosoma 21, compararla con la de otros cromosomas, etc.