Objetivo:
Determinar si existen genes asociados a la resistencia a antibioticos en el ensamblado
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA313047
(313047[BioProject]) AND Staphylococcus aureus
Solución
1.Generar una base de datos local para Blast
makeblastdb -in a26_1000.fa -parse_seqids -title "26_1000" -out a26_1000 -dbtype nucl
2. Descargar genes de resistencia en el servidor
3. Llevar a cabo una busqueda mediante un Blast local
blastn -query SA_resistance_genes.txt -db a26_1000 -max_target_seqs 1 -outfmt 6 -evalue 1E-5
Trabajar sobre el resultado del blast
Problema 1: Los identificadores no permiten identificar el gen ‘directamente’
Solución: Convertir las IDs (como NG_048135.1) a nombres de genes
Métodos:
- NCBI gene
- ID-mapping tool de UniProt
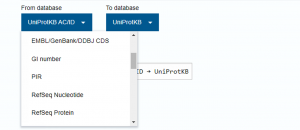
Para usar el ID-mapping de UniProt, necesitamos una lista de IDs.
Mediante el comando cut en linux podemos extraer una columna
cut -f 1 SA_resistance_genes_blastn.txt
indicando -f la columna que queremos extraer (en este caso la primera)
La lista la introducimos en el cuadro de texto de la página ID-mapping
Seleccionamos como ‘From database’ RefSeq Nucleotide
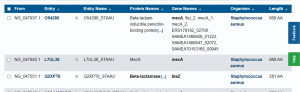
Obtendremos